Couplage entre la traduction et la dégradation des ARNm chez les eucaryotes
La synthèse des protéines par le ribosome à partir des ARN messagers (ARNm) est un processus finement régulé qui permet aux cellules de s’adapter pendant le cycle cellulaire ou en réponse à des facteurs extérieurs. A l’image de la transcription et de la traduction, la dégradation des ARNm eucaryotes apparaît maintenant comme un mécanisme très régulé qui permet aux cellules de moduler rapidement la production de protéines. Cela implique une forte connexion entre les machineries de traduction et de dégradation de ces ARNm. L’objectif des recherches conduites dans le groupe de Marc Graille est d’améliorer les connaissances actuelles sur les mécanismes de traduction et de dégradation des ARNm eucaryotes ainsi que leur dynamisme en utilisant des approches expérimentales variées : génétique de la levure, biochimie, biophysique, biologie structurale, …

Les thématiques développées dans le groupe sont :
Modifications post-transcriptionnelles et post-traductionnelles des facteurs (ARN ou protéines) impliquées dans la traduction.
La traduction est un processus à plusieurs étapes permettant la synthèse de protéines à partir des ARNm. Elle est réalisée par le ribosome, un complexe macromoléculaire composé de nombreuses protéines et d’ARN ribosomiques (ARNr), avec l’aide des ARN de transfert (ARNt) et des facteurs de traduction. Ces différents composants subissent des maturations post-transcriptionnelles et post-traductionnelles qui influencent l’efficacité et l’exactitude de la synthèse des ribosomes et/ou de la traduction avec des effets avérés sur la progression du cycle cellulaire. Plus de détails …
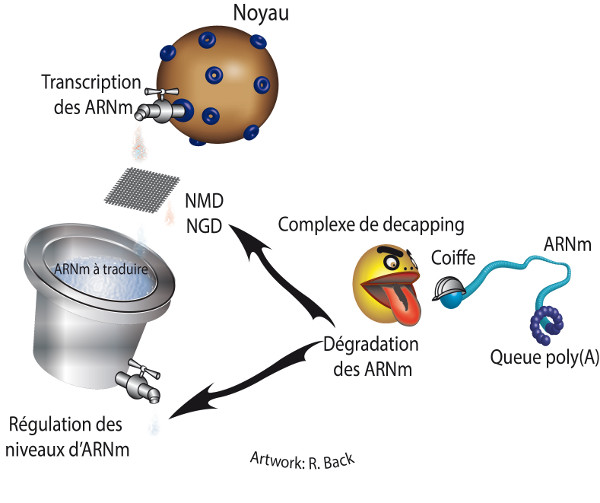
Les mécanismes de dégradation des ARNm.
La régulation des vitesses de dégradation des ARNm est un point de contrôle très important pour la détermination de l’abondance des transcrits cellulaires et donc des protéines à produire.
Les ARNm eucaryotes prêts à être traduits sont protégés d’une dégradation rapide et non contrôlée par deux éléments stabilisants en cis : une coiffe methylguanosine et une queue poly(A) aux extrémités 5’ et 3’, respectivement. En conséquence, la dégradation des ARNm nécessite un remodelage des particules ribonucléoprotéiques (mRNP) qui est déclenché par des signaux spécifiques et permet le recrutement d’activateurs de la dégradation et d’enzymes. Typiquement, la dégradation des ARNm eucaryotes est initiée par la dégradation de la queue poly(A) (ou déadénylation), suivie par l’élimination de la coiffe (ou decapping) protégeant l’extrémité 5’ des ARNm. Ensuite, les ARNm sont dégradés très rapidement dans le sens 5’—>3’ par l’exoribonucléase cytoplasmique Xrn1.
Le couplage entre la traduction et la dégradation des ARNm intervient aussi dans certains mécanismes de contrôle-qualité dont le rôle est de détecter et de dégrader les ARNm aberrants contenant des codons stop précoces (voie du Nonsense-Mediated mRNA Decay ou NMD) ou ceux bloquant les ribosomes lors de l’étape d’élongation (voie du No-Go Decay ou NGD) ainsi que les petites sous-unités du ribosome qui sont immatures ou inactives et bloquent donc l’étape d’élongation (voie du nonfunctional 18S-rRNA decay ou 18S-NRD)
(Graille & Séraphin ; Nature Reviews Mol Cell Biol ; 2012 / Fourati and Graille ; 2014). Plus de détails …