Les mécanismes de dégradation des ARNm.
a) Le decapping des ARNm.
Le decapping consiste en l’élimination de la coiffe protégeant l’extrémité 5’ des ARNm et est considérée comme une étape cruciale et irréversible avant la dégradation rapide des ARNm. Le decapping est réalisé suite au recrutement d’un complexe de l’enzyme Dcp2 et de son activateur Dcp1. Ce complexe a une faible activité intrinsèque et nécessite plusieurs facteurs accessoires pour être très efficace. Parmi ceux-ci, le complexe Lsm1-Lsm7 qui se fixe à l’extrémité 3’ des ARNm déadénylés, favorise le decapping et le recrutement de Pat1. Dhh1 et les protéines Edc1-3 (Enhancer of Decapping) favorisent aussi le decapping. Enfin, Dhh1, Scd6 et Pat1 agiraient comme des répresseur de la traduction en limitant la formation du complexe de pré-initiation 48S.
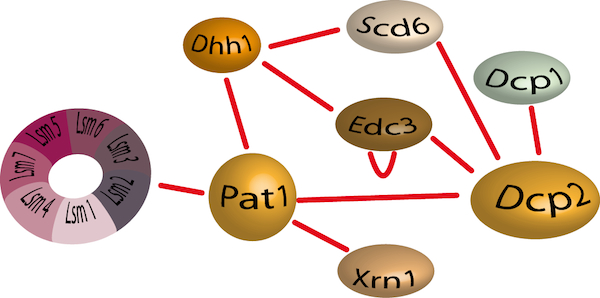
Le réseau d'interaction du complexe de décapping
Nous avons récemment résolu la structure cristallographique du domaine C-terminal de Pat1 de S. cerevisiae (Fourati et al; 2014; Plos One), une protéine échafaudage responsable du recrutement soit de répresseurs de la traduction soit d’activateurs de l’activité decapping, et identifié la région d’Edc3 responsable de l’interaction avec la protéine ribosomique Rps28a (Kolesnikova et al; 2013; NAR). Cependant, le mécanisme d’activation du decapping reste très largement inconnu. De façon intéressante, toutes ces protéines font parties d’assemblages protéiques dynamiques et multifonctionnels and l’importance de ces interactions protéine-protéine devient de plus en plus évident. Il est donc essentiel de comprendre les détails moléculaires responsables de la dynamique de ces complexes ainsi que les interactions impliquées, afin de décrire les mécanismes de dégradation des ARNm et la logique de leur régulation.
Collaborateur : Dr Bertrand Séraphin (IGBMC, CNRS, Univ. Strasbourg, Illkirch, France).
Nos articles sur cette thématique :
- Fourati Z; Kolesnikova O; Back R; Keller J; Charenton C; Taverniti V; Gaudon Plesse C; Lazar N; Durand D; van Tilbeurgh H; Séraphin B; Graille M. (2014) The C-terminal domain from S. cerevisiae Pat1 displays two conserved regions involved in decapping factor recruitment. Plos One, 9(5), e96828.
- Kolesnikova O; Back R; Graille M; Séraphin B. (2013) Identification of the Rps28 binding motif from yeast Edc3 involved in the autoregulatory feedback loop controlling RPS28B mRNA decay. Nucleic Acids Res, 41(20), 9514-9523.
b) La voie de dégradation des ARNm contenant des codons stop précoces (ou NMD).
On estime que les ARNm contenant des codons stop précoces (5-10% des ARNm eucaryotes) sont responsables d’1/3 des maladies génétiques héréditaires telles que la dystrophie musculaire de Duchenne (DMD) ou certaines formes de mucoviscidose ainsi que certains cancers. Cependant, les protéines tronquées synthétisées à partir de ces ARNm aberrants sont rarement détectés en raison de l’existence d’un mécanisme de surveillance: la voie de dégradation des ARNm contenant des codons stop précoces (ou NMD). La voie NMD détecte la plupart des ARNm aberrants et les détruit rapidement empêchant ainsi leur traduction. Parmi les protéines impliquées, figurent les facteurs de terminaison de la traduction erF1 et eRF3, les facteurs Upf1-3 spécifiques à la NMD et les protéines impliquées dans la dégradation des ARNm normaux.
Nous avons résolu la structure cristallographique du domaine N-terminal d’Upf2 et identifié une région très fortement conservée au niveau de ce domaine qui est cruciale pour la NMD, probablement via le recrutement d’un partenaire protéique (Fourati et al; 2014; J. Mol. Biol / Fourati and Graille; 2014).
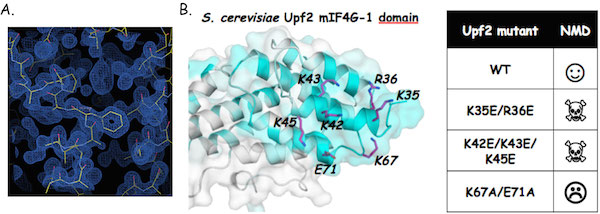
Le domaine N-terminal d’Upf2 est nécessaire pour la NMD
A. Carte de densité électronique expérimentale à 1.45Å de résolution obtenue grâce au programme ARCIMBOLDO (Courtesy from I. Uson). B. Des résidus très conservés au niveau du domaine N-terminal d’Upf2 sont essentiel pour la NMD.
Collaborateurs : Pr Allan Jacobson (Univ. Massachusetts Medical School, Worcester, USA) & Pr Mans Ehrenberg (Uppsala Univ., Sweden)
Nos articles sur cette thématique :
- Fourati Z; Roy B; Millan C; Coureux PD; Kervestin S; van Tilbeurgh H; He F; Uson I; Jacobson A; Graille M. (2014) A highly conserved region essential for NMD in the Upf2 N-terminal domain. J Mol. Biol. 426(22), 3689-3702.
- Fourati Z & Graille M. (2014) Cytoplasmic mRNA surveillance pathways. In « Fungal RNA biology » (Springer International Publishing). Editors : A. Sesma and T. von der Haar. ISBN 978-3-319-05687-6.
c) Mécanismes de contrôlée-qualité dégradant les ARN induisant des arrêts prolongés de la traudciton: les voies NGD, NSD et 18S-NRD.
En plus de la NMD, les cellules eucaryotes possèdent des mécanismes de surveillance dédiés à la dégradation des ARN induisant de fortes pauses des ribosomes lors de l’élongation de la traduction. Il s’agit des voies NGD (No-Go Decay), 18S-NRD (18S Nonfunctional rRNA decay) et NSD (Non-Stop Decay). Elles nécessitent les protéines Dom34 et Hbs1, qui sont structuralement similaires à eRF1 et eRF3, respectivement.
Pour comprendre le rôle de ces deux protéines dans ces mécanismes de surveillance, nous avons résolu les structures cristallines des protéines Dom34 et Hbs1 de levure et obtenu un modèle à basse résolution du complexe Dom34-Hbs1 par diffusion des rayons X aux petits angles (SAXS). Des expériences in vivo chez la levure S. cerevisiae ont montré que la fixation du nucléotide GTP par Hbs1 est nécessaire à la fois pour la NGD et pour la 18S-NRD. En revanche, nous avons aussi montré que l’interaction entre Dom34 et Hbs1 est nécessaire pour la voie NGD mais pas pour la voie 18S-NRD (Graille et al; 2008; JBC / van den Elzen ; 2010; Nat. Struct. Mol. Biol. / Graille & Séraphin; 2012; Nat. Rev. Mol. Cell. Biol./ Fourati and Graille; 2014).
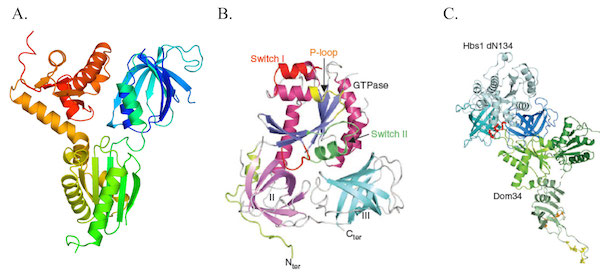
Le complexe Dom34-Hbs1.
Représentation des structures cristallines de Dom34 (A; colorée du bleu (N-terminus) au rouge (C-terminus) et, d’Hbs1 (B) ainsi que du modèle du complexe Hbs1-Dom34 obtenue par SAXS (C).
Collaborateur : Dr Bertrand Séraphin (IGBMC, CNRS, Univ. Strasbourg, Illkirch, France).
Nos articles sur cette thématique :
- Fourati Z & Graille M. (2014) Cytoplasmic mRNA surveillance pathways. In « Fungal RNA biology » (Springer International Publishing). Editors : A. Sesma and T. von der Haar. ISBN 978-3-319-05687-6.
- Graille M & Séraphin B. (2012) Surveillance pathways rescuing eukaryotic ribosomes lost in translation. Nat. Rev. Mol. Cell Biol., 13, 727-735 (Review).
- van den Elzen AMG, Henri J, Lazar N, Gas-Lopez M, Durand D, Lacroute F, NicaiseM, van Tilbeurgh H, Séraphin B, Graille M. (2010) Dissection of Dom34-Hbs1 reveals independent functions in two RNA quality control pathways. Nat. Struct. Mol. Biol. 17(12), 1446-53.
- Graille M, Chaillet M, van Tilbeurgh H. (2008) Structure of yeast Dom34: a protein related to translation termination factor eRF1 and involved in No-Go decay. J Biol Chem. 283(11), 7145-54.


