Modifications post-transcriptionnelles et post-traductionnelles des facteurs (ARN ou protéines) impliquées dans la traduction
a) Trm112, un acteur central impliqué dans la maturation de plusieurs facteurs intervenant dans la traduction.
Trm112, une petite protéine à doigt de zinc de 15 kDa, interagit avec et active 4 méthyltransférases (MTases) : Mtq2, Trm9, Trm11 et Bud23, qui modifient des facteurs impliqués dans la synthèse des protéines.
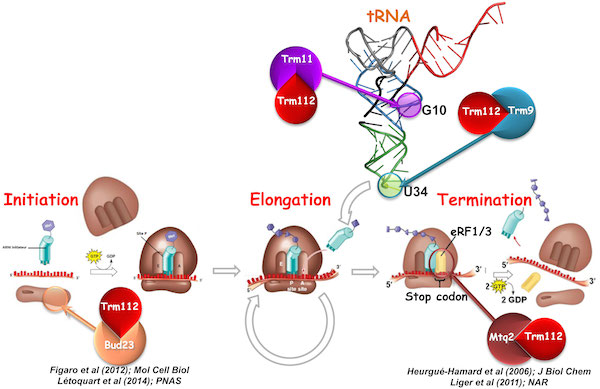
Le complexe Mtq2-Trm112 méthyle le facteur de terminaison de la traduction eRF1 sur la chaine latérale de la glutamine du motif universellement conservé GGQ qui est directement impliqué nda la libération des protéines nouvellement synthétisées (Heurgué-Hamard et al ; 2006 ; JBC / Liger et al ; 2011 ; NAR). Le complexe Bud23-Trm112 intervient dans la biogenèse du ribosome en méthylant la guanosine 1575 de l’ARNr 18S (Figaro et al ; 2012 ; Mol Cell Biol / Létoquart et al ; 2014 ; PNAS). Les complexes Trm11-Trm112 et Trm9-Trm112 modifient les ARNt. Trm112 joue donc un rôle unique comme plate-forme activatrice de 4 MTases impliquées dans la modification des ARNr, ARNt et de facteurs de la traduction, la plaçant à l’interface entre la synthèse du ribosome et sa fonction. Chez l’Homme, certaines de ces protéines sont impliquées dans des maladies telles que certains cancers.
Notre objectif est de caractériser les mécanismes utilisés par ces enzymes pour reconnaître et modifier leurs substrats afin de mieux comprendre leur rôles biologiques et la fonction des modifications qu’elles réalisent.

La structure cristallographique du complexe Bud23-Trm112 de S. cerevisiae. La superposition du site actif de Bud23 avec celui de la xanthosine MTase de Coffea canéphore a permis d’identifier les résidus catalytiques et de révéler que le complexe Bud23-Trm112 ne peut pas fixer une sous-unité 40S mature, impliquant que ce complexe est recruté au niveau des précurseurs des ribosomes lors de la synthèse de la 40S. Image réalisée par Gabrielle Bourgeois.
Collaborateurs : Dr Valérie Heurgué-Hamard (IBPC, CNRS, Paris, France) & Pr Denis L.J. Lafontaine (Université Libre de Bruxelles, Charleroi, Belgium).
Nos articles sur cette thématique :
- Létoquart J, Tran N.V, Caroline V, Alexandrov A, Lazar N, van Tilbeurgh H, Liger D, Graille M. (2015) Insights into molecular plasticity in protein complexes from Trm9-Trm112 tRNA modifying enzyme crystal structure. Nucleic Acids Res; 43(22), 10989-11002.
- Létoquart J ; Huvelle E ; Wacheul L ; Bourgeois G ; Zorbas C ; Graille M ; Heurgué-Hamard V ; Lafontaine D.L.J. (2014) Structural and functional studies of Bud23-Trm112 reveal 18S rRNA N7-G1575 methylation occurs on late 40S precursor ribosomes. Proc Natl Acad Sci U S A. 111(51), E5518-26.
- Figaro S, Wacheul L, Schillewaert S, Graille M, Huvelle E, Mongeard R, Zorbas C, Lafontaine DLJ, Heurgué-Hamard. (2012) Trm112 is required for Bud23-mediated methylation of the 18S rRNA at position G1575. Mol. Cell. Biol. 32(12), 2254-67.
- Graille M, Figaro S, Kervestin, S, Buckingham RH, Liger D, Heurgué-Hamard V. (2012) Methylation of class I translation termination factors : structural and functional aspects. Biochimie. 94, 1533-43 (Review).
- Liger D, Mora L, Lazar N, Figaro S, Henri J, Scrima N, Buckingham RH, van Tilbeurgh H, Heurgué-Hamard V, Graille M. (2011) Mechanism of activation of methyltransferases involved in translation by the Trm112 « hub » protein. Nucleic Acids Res. 39(14), 6249-59.
- Heurgué-Hamard V, Graille M, Scrima N, Ulryck N, Champ S, van Tilbeurgh H, Buckingham RH. (2006) The zinc finger protein Ynr046w is plurifunctional and a component of the eRF1 methyltransferase in yeast. J Biol Chem. 281(47), 36140-8.
- Graille M, Heurgue-Hamard V, Champ S, Mora L, Scrima N, Ulryck N, van Tilbeurgh H, Buckingham RH. (2005) Molecular Basis for Bacterial Class I Release Factor Methylation by PrmC. Mol. Cell. 20(6), 917-27.
b) La prolyl-hydroxylase Tpa1 et son partenaire Ett1 régulent l’efficacité de terminaison de la traduction
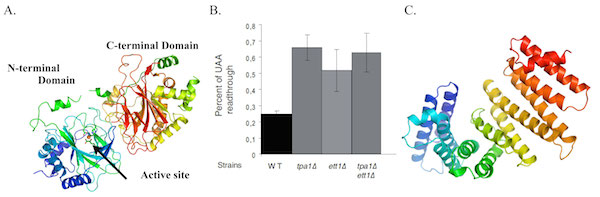
La protéine Tpa1 a été identifiée chez la levure S. cerevisiae yeast comme étant un composant (avec eRF1, eRF3 et Pab1) d’une mRNP associée à l’extrémité 3’ des ARNm et il a été montré qu’elle influence la reconnaissance des codons stop.
Pour étudier plus en détail sa fonction, nous avons résolu la structure cristalline de la protéine Tpa1 de S. cerevisiae, révélant une forte similarité structurale avec les prolyl-4-hydroxylases (enzymes catalysant l’hydroxylation des chaines latérales de résidus proline d’une manière dépendant de l’O2. Nous avons également identifié la protéine Ett1 comme nouveau facteur influençant reconnaissance des codons stop (Henri et al ; 2010 ; JBC). Il a été récemment montré que Tpa1 est effectivement une prolyl hydroxylase modifiant une proline conservée d’une protéine du ribosome. En parallèle, nous avons résolu la structure cristalline de la protéine Nro1, l’orthologue d’Ett1 chez la levure S. pombe, et montré que cette protéine intervient dans le même mécanisme que Tpa1 (Rispal et al ; 2011 ; RNA).
Collaborateurs : Dr Bertrand Séraphin (IGBMC, CNRS, Univ. Strasbourg, Illkirch, France).
Nos articles sur cette thématique :
- Henri J, Rispal D, Bayart E, van Tilbeurgh H, Séraphin B, Graille M. (2010) Structural and functional insights into S. cerevisiae Tpa1, a putative prolyl hydroxylase influencing translation termination and transcription. J Biol Chem, 285(40), 30767-78.
- Rispal D, Henri J, van Tilbeurgh H, Graille M, Séraphin B. (2011) Structural and functional analysis of Nro1/Ett1 : a protein involved in translation termination in S. cerevisiae and in O2-mediated gene control in S. pombe. RNA. 17(7), 1213-24.


