Comment extraire toute l'information cachée dans la trajectoire d'une biomolécule ?
Le marquage de biomolécules par des fluorophores organiques ou inorganiques permet de détecter leur localisation dans les cellules.Grâce au suivi dynamique des biomolécules individuelles marquées, il est possible de reconstruire leur trajectoire et d'obtenir des informations précieuses sur leur fonctionnement. Dans la membrane cellulaire, en particulier, selon le type de biomolécule, des mouvements libres (browniens) ou confinés à l'intérieur de compartiments membranaires sont observés. Des modèles différents sont proposés pour expliquer l'origine de ces compartiments et les trajectoires mesurées devraient permettre de trancher entre ces modèles. Cependant les techniques d'analyse actuelles n'exploitent qu'une petite partie des informations cachées dans ces trajectoires.Des chercheurs de l'Institut Pasteur (URA 2171 CNRS/Unité Pasteur Génétique in Silico et Unité Bactéries Anaérobies et Toxines) et duLaboratoire d'Optique et Biosciences (UMR 7645 CNRS/ U696 INSERM/ Ecole Polytechnique) viennent de proposer une technique d'inférence bayésienne qui exploite l'ensemble de l'information contenue dans ce type de trajectoire et de l'appliquer pour extraire les forces subies par le récepteur d'une toxine à l'intérieur d'un compartiment membranaire.
En théorie des probabilités et en statistique, l'inférence bayésienne détermine la probabilité d'avoir une certaine valeur d'un paramètre étant donnée une observation qui dépend de ce paramètre. Elle est utilisée dans des domaines trèsvariés, entre autres pour calculer la probabilité de « faux positifs »pour des tests médicaux. Les équipes de l'Institut Pasteur et de l'Ecole Polytechnique ont montré que cette approche pouvait également être appliquée à l'analyse des trajectoires de biomolécules. Elle a d'abord été validée sur des trajectoires calculées numériquement et ensuite utilisée pour déterminer les forces qui s'exercent sur les récepteurs d'une toxine se trouvant confinés dans des compartiments membranaires. Des trajectoires continues très longues ont été obtenues grâce à un marquage par des nanoparticules d'ions lanthanides qui ne présentent pas de clignotement (synthétisées au Laboratoire de Physique de la Matière Condensée, UMR7643 CNRS/Ecole Polytechnique). Cette nouvelle technique est cependant applicable de façon générale à tout type de marquage et de biomolécule et sera précieuse pour la compréhension des mécanismes de confinement dans les membranes cellulaires.
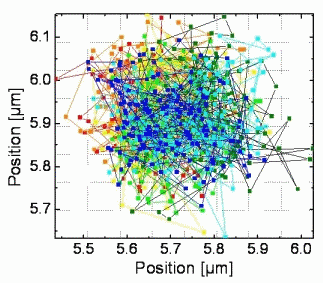
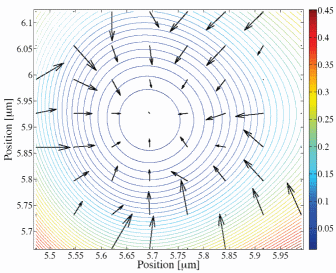
Trajectoire d'un récepteur membranaire (gauche) et champs de forces etde potentiel ressentis par le récepteur (droite) extraits avec la technique d'inférence.La longueur des flèches est proportionnelle à l'amplitude de la force. Le potentiel est indiqué par des courbes de niveaux.
Contacts
Ecole Polytechnique, Laboratoire d'Optique et Biosciences : Antigoni Alexandrou, antigoni.alexandrou at polytechnique.edu
Institut Pasteur, Unité génétique in silico : Jean-Baptiste Masson, jbmasson at pasteur.fr
J.-B. Masson, D. Casanova, S. Türkcan, G. Voisinne, M. R. Popoff, M. Vergassola, A. Alexandrou
Inferring Maps of Forces inside Cell Membrane Microdomains Phys. Rev. Lett. 102, 048103 (2009)
